Alphafold 3: révolutionner la structure moléculaire et la prédiction d'interaction
Depuis son lancement en 2020, plus de 2 millions de chercheurs ont utilisé AlphaFold 2 de Google DeepMind pour prédire les structures des protéines, contribuant à des avancées comme le développement de vaccins et les traitements contre le cancer. Ce modèle a relevé un défi qui a déconcerté les scientifiques pendant plus d’un demi-siècle. Mais l’équipe de Google DeepMind ne s’est pas arrêtée là ; au lieu de se reposer sur leurs lauriers, ils ont retroussé leurs manches et ont commencé à travailler sur AlphaFold 3.
Lancé en mai par Google DeepMind et Isomorphic Labs, AlphaFold 3 passe à la vitesse supérieure. Il ne se contente pas de prédire le repliement des protéines ; il prévoit également la structure et les interactions de toutes sortes de molécules de la vie, y compris l’ADN, l’ARN et les ligands — ces petites molécules qui se fixent aux protéines.
« Avec AlphaFold 2, nous avons fait d’énormes progrès dans la résolution du puzzle du repliement des protéines, mais la communauté scientifique est passée à des sujets plus complexes », explique Jonas Adler, scientifique de recherche chez Google DeepMind. « Les chercheurs se penchent maintenant sur des détails comme la liaison des petites molécules ou le fonctionnement de l’ARN, des domaines où AlphaFold 2 était limité. Pour suivre les dernières avancées en biologie et chimie, nous avions besoin d’un outil capable de gérer toutes sortes de biomolécules. »
« Tout » ici inclut les ligands, cruciaux car ils constituent environ la moitié de tous les médicaments. « Chez Isomorphic Labs, nous voyons l’énorme potentiel d’AlphaFold 3 pour concevoir des médicaments de manière rationnelle, et nous l’utilisons déjà tous les jours », déclare Adrian Stecula, responsable de recherche chez Isomorphic Labs. « Nous explorons comment de nouvelles petites molécules se lient à de nouvelles cibles médicitiques, comment les protéines interagissent avec l’ADN et l’ARN, et comment des ajustements chimiques affectent les structures des protéines — le nouveau modèle ouvre toutes ces possibilités. »
Incorporer ces types de molécules supplémentaires signifiait gérer bien plus de combinaisons. « Les protéines sont assez simples ; il n’y a que 20 acides aminés standards », note Jonas. « Mais les petites molécules ? Elles sont très variées, avec des possibilités infinies. Elles sont extrêmement diverses. »
Constatant qu’une base de données exhaustive était irréalisable, l’équipe a lancé AlphaFold Server. Cet outil gratuit permet aux scientifiques de saisir leurs propres séquences, et AlphaFold génère les complexes moléculaires pour eux. Depuis son lancement en mai, il a été utilisé pour créer plus d’un million de structures.
« C’est comme Google Maps, mais pour les complexes moléculaires », explique Lindsay Willmore, ingénieur de recherche chez Google DeepMind. « Même si vous n’êtes pas codeur, vous pouvez simplement copier et coller vos séquences de protéines, d’ADN, d’ARN ou de petites molécules, cliquer sur un bouton et attendre quelques minutes. Vous obtiendrez votre structure avec des métriques de confiance pour évaluer la prédiction. »
Pour qu’AlphaFold 3 fonctionne avec une si large gamme de biomolécules, l’équipe a élargi les données d’entraînement pour inclure l’ADN, l’ARN, les petites molécules, et plus encore. « Nous nous sommes dit : ‘Pourquoi ne pas entraîner sur tout ce que nous avons dans notre ensemble de données qui nous a aidés avec les protéines et voir jusqu’où nous pouvons aller ?’ », explique Lindsay. « Il s’avère que nous sommes allés assez loin. »
Un changement clé dans AlphaFold 3 concerne la dernière partie du modèle qui crée la structure. Alors qu’AlphaFold 2 utilisait un module complexe basé sur la géométrie, AlphaFold 3 est passé à un modèle génératif plus simple basé sur la diffusion, similaire à d’autres modèles avancés de génération d’images comme Imagen. Ce changement a simplifié la gestion des nouveaux types de molécules.
Cependant, ce changement a apporté un nouveau défi : le modèle de diffusion créait par erreur une structure « ordonnée » pour des régions protéiques qui sont en réalité « désordonnées » — imaginez essayer d’organiser un tas de spaghettis chaotiques en une spirale nette.
Ainsi, l’équipe s’est tournée vers AlphaFold 2, qui excelle dans la prédiction de ces interactions désordonnées. « Nous avons utilisé ces prédictions d’AlphaFold 2 pour entraîner AlphaFold 3, lui apprenant à reconnaître et prédire le désordre », explique Lindsay.
« Nous avons un dicton : ‘Faites confiance aux fusilli, rejetez les spaghettis’ », ajoute Jonas en riant.
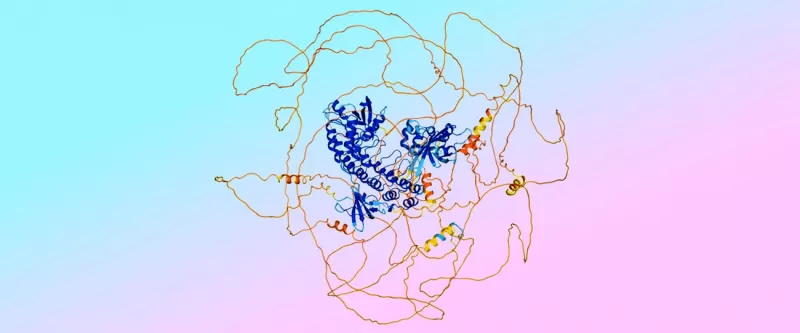
Un exemple de prédiction d’AlphaFold 3, montrant des régions « fusilli » ordonnées en bleu et des régions « spaghettis » désordonnées en orange. Les couleurs indiquent la confiance du modèle dans l’exactitude de la prédiction.
L’équipe est enthousiaste à l’idée de voir comment AlphaFold 3 sera utilisé dans des domaines comme la génomique et la conception de médicaments. « C’est incroyable de voir jusqu’où nous sommes allés », dit Jonas. « Ce qui était autrefois difficile est maintenant facile, et ce qui était impossible est désormais à portée de main. Il reste encore des défis à relever, mais nous sommes ravis de ce qu’AlphaFold 3 peut nous aider à accomplir. »
 "Dot AI Companion Appounces Closure, Discontinues Personalized Service" (L'application Dot AI Companion annonce sa fermeture et interrompt son service personnalisé)
Dot, une application d'IA conçue pour servir d'ami personnel et de confident, va cesser ses activités, selon une annonce faite vendredi par ses développeurs. New Computer, la startup à l'origine de Do
"Dot AI Companion Appounces Closure, Discontinues Personalized Service" (L'application Dot AI Companion annonce sa fermeture et interrompt son service personnalisé)
Dot, une application d'IA conçue pour servir d'ami personnel et de confident, va cesser ses activités, selon une annonce faite vendredi par ses développeurs. New Computer, la startup à l'origine de Do
 Anthropic résout une affaire de piratage de livres générés par l'IA
Anthropic a trouvé une solution à un important litige en matière de droits d'auteur avec des auteurs américains, en acceptant une proposition de règlement de recours collectif qui évite un procès pote
Anthropic résout une affaire de piratage de livres générés par l'IA
Anthropic a trouvé une solution à un important litige en matière de droits d'auteur avec des auteurs américains, en acceptant une proposition de règlement de recours collectif qui évite un procès pote
 Figma met à la disposition de tous les utilisateurs un outil de création d'applications basé sur l'IA
Figma Make, la plateforme de développement innovante de type prompt-to-app dévoilée au début de l'année, est officiellement sortie de la phase bêta et a été déployée pour tous les utilisateurs. Cet ou
commentaires (21)
0/200
Figma met à la disposition de tous les utilisateurs un outil de création d'applications basé sur l'IA
Figma Make, la plateforme de développement innovante de type prompt-to-app dévoilée au début de l'année, est officiellement sortie de la phase bêta et a été déployée pour tous les utilisateurs. Cet ou
commentaires (21)
0/200
![HarryClark]() HarryClark
HarryClark
 21 août 2025 09:01:19 UTC+02:00
21 août 2025 09:01:19 UTC+02:00
AlphaFold 3 sounds like a game-changer! Predicting molecular interactions could speed up drug discovery big time. Curious how it stacks up against traditional lab methods. 🧬


 0
0
![PeterThomas]() PeterThomas
PeterThomas
 17 août 2025 01:01:00 UTC+02:00
17 août 2025 01:01:00 UTC+02:00
AlphaFold 3 sounds like a game-changer! Predicting molecular interactions with such precision could really speed up drug discovery. I'm curious how it stacks up against traditional lab methods in terms of cost and time. Anyone know? 🤔


 0
0
![JoseLewis]() JoseLewis
JoseLewis
 14 août 2025 13:00:59 UTC+02:00
14 août 2025 13:00:59 UTC+02:00
AlphaFold 3 sounds like a game-changer! Predicting molecular interactions could speed up drug discovery big time. Excited to see how it shapes medical breakthroughs! 🚀


 0
0
![ElijahCollins]() ElijahCollins
ElijahCollins
 14 août 2025 05:01:00 UTC+02:00
14 août 2025 05:01:00 UTC+02:00
AlphaFold 3 sounds like a game-changer! Predicting molecular interactions could speed up drug discovery big time. Curious how it stacks against human researchers—will it outsmart us or just make our coffee breaks longer? 😄


 0
0
![GregorySmith]() GregorySmith
GregorySmith
 6 août 2025 04:01:00 UTC+02:00
6 août 2025 04:01:00 UTC+02:00
AlphaFold 3 sounds like a game-changer! Predicting molecular interactions with such precision could unlock new frontiers in medicine. But I wonder, will this tech stay accessible to all researchers, or get locked behind paywalls? 🤔


 0
0
![BruceThomas]() BruceThomas
BruceThomas
 1 août 2025 08:08:50 UTC+02:00
1 août 2025 08:08:50 UTC+02:00
AlphaFold 3 sounds like a game-changer for science! Predicting molecular interactions could speed up drug discovery big time. Excited to see where this goes! 🚀


 0
0

Depuis son lancement en 2020, plus de 2 millions de chercheurs ont utilisé AlphaFold 2 de Google DeepMind pour prédire les structures des protéines, contribuant à des avancées comme le développement de vaccins et les traitements contre le cancer. Ce modèle a relevé un défi qui a déconcerté les scientifiques pendant plus d’un demi-siècle. Mais l’équipe de Google DeepMind ne s’est pas arrêtée là ; au lieu de se reposer sur leurs lauriers, ils ont retroussé leurs manches et ont commencé à travailler sur AlphaFold 3.
Lancé en mai par Google DeepMind et Isomorphic Labs, AlphaFold 3 passe à la vitesse supérieure. Il ne se contente pas de prédire le repliement des protéines ; il prévoit également la structure et les interactions de toutes sortes de molécules de la vie, y compris l’ADN, l’ARN et les ligands — ces petites molécules qui se fixent aux protéines.
« Avec AlphaFold 2, nous avons fait d’énormes progrès dans la résolution du puzzle du repliement des protéines, mais la communauté scientifique est passée à des sujets plus complexes », explique Jonas Adler, scientifique de recherche chez Google DeepMind. « Les chercheurs se penchent maintenant sur des détails comme la liaison des petites molécules ou le fonctionnement de l’ARN, des domaines où AlphaFold 2 était limité. Pour suivre les dernières avancées en biologie et chimie, nous avions besoin d’un outil capable de gérer toutes sortes de biomolécules. »
« Tout » ici inclut les ligands, cruciaux car ils constituent environ la moitié de tous les médicaments. « Chez Isomorphic Labs, nous voyons l’énorme potentiel d’AlphaFold 3 pour concevoir des médicaments de manière rationnelle, et nous l’utilisons déjà tous les jours », déclare Adrian Stecula, responsable de recherche chez Isomorphic Labs. « Nous explorons comment de nouvelles petites molécules se lient à de nouvelles cibles médicitiques, comment les protéines interagissent avec l’ADN et l’ARN, et comment des ajustements chimiques affectent les structures des protéines — le nouveau modèle ouvre toutes ces possibilités. »
Incorporer ces types de molécules supplémentaires signifiait gérer bien plus de combinaisons. « Les protéines sont assez simples ; il n’y a que 20 acides aminés standards », note Jonas. « Mais les petites molécules ? Elles sont très variées, avec des possibilités infinies. Elles sont extrêmement diverses. »
« C’est comme Google Maps, mais pour les complexes moléculaires », explique Lindsay Willmore, ingénieur de recherche chez Google DeepMind. « Même si vous n’êtes pas codeur, vous pouvez simplement copier et coller vos séquences de protéines, d’ADN, d’ARN ou de petites molécules, cliquer sur un bouton et attendre quelques minutes. Vous obtiendrez votre structure avec des métriques de confiance pour évaluer la prédiction. »
Pour qu’AlphaFold 3 fonctionne avec une si large gamme de biomolécules, l’équipe a élargi les données d’entraînement pour inclure l’ADN, l’ARN, les petites molécules, et plus encore. « Nous nous sommes dit : ‘Pourquoi ne pas entraîner sur tout ce que nous avons dans notre ensemble de données qui nous a aidés avec les protéines et voir jusqu’où nous pouvons aller ?’ », explique Lindsay. « Il s’avère que nous sommes allés assez loin. »
Un changement clé dans AlphaFold 3 concerne la dernière partie du modèle qui crée la structure. Alors qu’AlphaFold 2 utilisait un module complexe basé sur la géométrie, AlphaFold 3 est passé à un modèle génératif plus simple basé sur la diffusion, similaire à d’autres modèles avancés de génération d’images comme Imagen. Ce changement a simplifié la gestion des nouveaux types de molécules.
Cependant, ce changement a apporté un nouveau défi : le modèle de diffusion créait par erreur une structure « ordonnée » pour des régions protéiques qui sont en réalité « désordonnées » — imaginez essayer d’organiser un tas de spaghettis chaotiques en une spirale nette.
Ainsi, l’équipe s’est tournée vers AlphaFold 2, qui excelle dans la prédiction de ces interactions désordonnées. « Nous avons utilisé ces prédictions d’AlphaFold 2 pour entraîner AlphaFold 3, lui apprenant à reconnaître et prédire le désordre », explique Lindsay.
« Nous avons un dicton : ‘Faites confiance aux fusilli, rejetez les spaghettis’ », ajoute Jonas en riant.
L’équipe est enthousiaste à l’idée de voir comment AlphaFold 3 sera utilisé dans des domaines comme la génomique et la conception de médicaments. « C’est incroyable de voir jusqu’où nous sommes allés », dit Jonas. « Ce qui était autrefois difficile est maintenant facile, et ce qui était impossible est désormais à portée de main. Il reste encore des défis à relever, mais nous sommes ravis de ce qu’AlphaFold 3 peut nous aider à accomplir. »
 Anthropic résout une affaire de piratage de livres générés par l'IA
Anthropic a trouvé une solution à un important litige en matière de droits d'auteur avec des auteurs américains, en acceptant une proposition de règlement de recours collectif qui évite un procès pote
Anthropic résout une affaire de piratage de livres générés par l'IA
Anthropic a trouvé une solution à un important litige en matière de droits d'auteur avec des auteurs américains, en acceptant une proposition de règlement de recours collectif qui évite un procès pote
 Figma met à la disposition de tous les utilisateurs un outil de création d'applications basé sur l'IA
Figma Make, la plateforme de développement innovante de type prompt-to-app dévoilée au début de l'année, est officiellement sortie de la phase bêta et a été déployée pour tous les utilisateurs. Cet ou
Figma met à la disposition de tous les utilisateurs un outil de création d'applications basé sur l'IA
Figma Make, la plateforme de développement innovante de type prompt-to-app dévoilée au début de l'année, est officiellement sortie de la phase bêta et a été déployée pour tous les utilisateurs. Cet ou
 21 août 2025 09:01:19 UTC+02:00
21 août 2025 09:01:19 UTC+02:00
AlphaFold 3 sounds like a game-changer! Predicting molecular interactions could speed up drug discovery big time. Curious how it stacks up against traditional lab methods. 🧬


 0
0
 17 août 2025 01:01:00 UTC+02:00
17 août 2025 01:01:00 UTC+02:00
AlphaFold 3 sounds like a game-changer! Predicting molecular interactions with such precision could really speed up drug discovery. I'm curious how it stacks up against traditional lab methods in terms of cost and time. Anyone know? 🤔


 0
0
 14 août 2025 13:00:59 UTC+02:00
14 août 2025 13:00:59 UTC+02:00
AlphaFold 3 sounds like a game-changer! Predicting molecular interactions could speed up drug discovery big time. Excited to see how it shapes medical breakthroughs! 🚀


 0
0
 14 août 2025 05:01:00 UTC+02:00
14 août 2025 05:01:00 UTC+02:00
AlphaFold 3 sounds like a game-changer! Predicting molecular interactions could speed up drug discovery big time. Curious how it stacks against human researchers—will it outsmart us or just make our coffee breaks longer? 😄


 0
0
 6 août 2025 04:01:00 UTC+02:00
6 août 2025 04:01:00 UTC+02:00
AlphaFold 3 sounds like a game-changer! Predicting molecular interactions with such precision could unlock new frontiers in medicine. But I wonder, will this tech stay accessible to all researchers, or get locked behind paywalls? 🤔


 0
0
 1 août 2025 08:08:50 UTC+02:00
1 août 2025 08:08:50 UTC+02:00
AlphaFold 3 sounds like a game-changer for science! Predicting molecular interactions could speed up drug discovery big time. Excited to see where this goes! 🚀


 0
0