Alphafold 3: revolutionieren molekulare Struktur und Interaktionsvorhersage
Seit der Einführung im Jahr 2020 haben über 2 Millionen Forscher Google DeepMinds AlphaFold 2 genutzt, um Proteinstrukturen vorherzusagen und Durchbrüche wie Impfstoffentwicklung und Krebsbehandlungen zu fördern. Dieses Modell löste ein Problem, das Wissenschaftler über ein halbes Jahrhundert lang beschäftigte. Doch das Team bei Google DeepMind ruhte sich nicht auf seinen Lorbeeren aus, sondern krempelte die Ärmel hoch und begann an AlphaFold 3 zu arbeiten.
Im Mai von Google DeepMind und Isomorphic Labs eingeführt, hebt AlphaFold 3 die Messlatte höher. Es sagt nicht nur die Proteinfaltung voraus, sondern prognostiziert auch die Struktur und Wechselwirkungen aller Arten von Molekülen des Lebens, einschließlich DNA, RNA und Liganden – jenen kleinen Molekülen, die an Proteine binden.
„Mit AlphaFold 2 haben wir große Fortschritte bei der Lösung des Proteinfaltungspuzzles gemacht, aber die wissenschaftliche Gemeinschaft ist zu komplexeren Themen übergegangen“, erklärt Jonas Adler, Forschungswissenschaftler bei Google DeepMind. „Forscher beschäftigen sich jetzt mit Details wie der Bindung kleiner Moleküle oder der Funktionsweise von RNA, Bereiche, in denen AlphaFold 2 nicht ausreichte. Um mit den neuesten Entwicklungen in Biologie und Chemie Schritt zu halten, brauchten wir ein Werkzeug, das alle Arten von Biomolekülen verarbeiten kann.“
„Alles“ umfasst hier Liganden, die entscheidend sind, da sie etwa die Hälfte aller Medikamente ausmachen. „Bei Isomorphic Labs sehen wir das enorme Potenzial von AlphaFold 3 für die rationale Arzneimittelentwicklung, und wir nutzen es bereits täglich“, sagt Adrian Stecula, Forschungsleiter bei Isomorphic Labs. „Wir erforschen, wie neue kleine Moleküle an neue Arzneimittelziele binden, wie Proteine mit DNA und RNA interagieren und wie chemische Veränderungen Proteinstrukturen beeinflussen – das neue Modell eröffnet all diese Möglichkeiten.“
Die Einbeziehung dieser zusätzlichen Molekültypen bedeutete, mit einer Vielzahl von Kombinationen umzugehen. „Proteine sind ziemlich einfach; es gibt nur 20 Standardaminosäuren“, bemerkt Jonas. „Aber kleine Moleküle? Die sind überall, mit unendlichen Möglichkeiten. Sie sind extrem vielfältig.“
Da eine umfassende Datenbank ausgeschlossen war, startete das Team den AlphaFold Server. Dieses kostenlose Tool ermöglicht es Wissenschaftlern, ihre eigenen Sequenzen einzugeben, und AlphaFold generiert die molekularen Komplexe für sie. Seit der Einführung im Mai wurden über 1 Million Strukturen erstellt.
„Es ist wie Google Maps, aber für molekulare Komplexe“, sagt Lindsay Willmore, Forschungsingenieurin bei Google DeepMind. „Auch wenn du kein Programmierer bist, kannst du einfach deine Protein-, DNA-, RNA- oder kleinen Molekülsequenzen kopieren und einfügen, auf einen Button klicken und ein paar Minuten warten. Du erhältst deine Struktur zusammen mit Konfidenzmetriken, um die Vorhersage zu bewerten.“
Um AlphaFold 3 mit einer so breiten Palette von Biomolekülen zu nutzen, erweiterte das Team die Trainingsdaten um DNA, RNA, kleine Moleküle und mehr. „Wir dachten: ‚Warum nicht auf allem in unserem Datensatz trainieren, was uns bei Proteinen geholfen hat, und sehen, wie weit wir kommen?‘“, erklärt Lindsay. „Es stellte sich heraus, dass wir ziemlich weit kamen.“
Eine wesentliche Änderung in AlphaFold 3 war im letzten Teil des Modells, das die Struktur erstellt. Während AlphaFold 2 ein komplexes, geometriebasiertes Modul verwendete, wechselte AlphaFold 3 zu einem einfacheren generativen Modell basierend auf Diffusion, ähnlich wie andere fortschrittliche Bildgenerierungsmodelle wie Imagen. Diese Änderung vereinfachte den Umgang mit den neuen Molekültypen.
Diese Umstellung brachte jedoch eine neue Herausforderung: Das Diffusionsmodell erstellte fälschlicherweise eine „geordnete“ Struktur für Proteinregionen, die tatsächlich „ungeordnet“ sind – stell dir vor, du versuchst, einen Haufen chaotischer Spaghetti in eine ordentliche Spirale zu bringen.
Also griff das Team auf AlphaFold 2 zurück, das bei der Vorhersage dieser ungeordneten Wechselwirkungen glänzt. „Wir haben diese Vorhersagen von AlphaFold 2 genutzt, um AlphaFold 3 zu trainieren und ihm beizubringen, Unordnung zu erkennen und vorherzusagen“, sagt Lindsay.
„Wir haben einen Spruch: ‚Vertraue den Fusilli, lehne die Spaghetti ab‘“, fügt Jonas lachend hinzu.
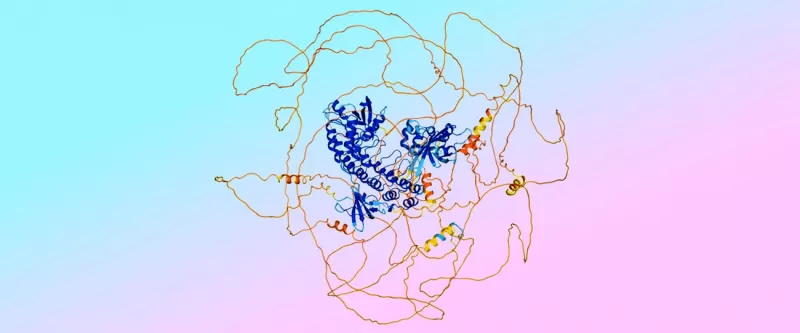
Ein Beispiel für eine Vorhersage von AlphaFold 3, das geordnete „Fusilli“-Regionen in Blau und ungeordnete „Spaghetti“-Regionen in Orange zeigt. Die Farben zeigen das Vertrauen des Modells in die Genauigkeit der Vorhersage.
Das Team ist begeistert, wie AlphaFold 3 in Bereichen wie Genomik und Arzneimitteldesign eingesetzt wird. „Es ist erstaunlich zu sehen, wie weit wir gekommen sind“, sagt Jonas. „Was einst schwierig war, ist jetzt einfach, und was unmöglich war, ist jetzt in Reichweite. Es gibt immer noch harte Nüsse zu knacken, aber wir sind begeistert, was AlphaFold 3 uns ermöglicht.“
 "Dot AI Companion App kündigt Schließung an, stellt personalisierten Service ein"
Dot, eine KI-Begleitanwendung, die als persönlicher Freund und Vertrauter fungieren soll, wird laut einer Ankündigung seiner Entwickler vom Freitag seinen Betrieb einstellen. New Computer, das Startup
"Dot AI Companion App kündigt Schließung an, stellt personalisierten Service ein"
Dot, eine KI-Begleitanwendung, die als persönlicher Freund und Vertrauter fungieren soll, wird laut einer Ankündigung seiner Entwickler vom Freitag seinen Betrieb einstellen. New Computer, das Startup
 Anthropic löst Rechtsstreit über AI-generierte Buchpiraterie
Anthropic hat einen bedeutenden Urheberrechtsstreit mit US-Autoren beigelegt. Das Unternehmen hat einer vorgeschlagenen Sammelklage zugestimmt, mit der ein potenziell kostspieliger Prozess vermieden w
Anthropic löst Rechtsstreit über AI-generierte Buchpiraterie
Anthropic hat einen bedeutenden Urheberrechtsstreit mit US-Autoren beigelegt. Das Unternehmen hat einer vorgeschlagenen Sammelklage zugestimmt, mit der ein potenziell kostspieliger Prozess vermieden w
 Figma veröffentlicht AI-gestütztes App Builder Tool für alle Benutzer
Figma Make, die innovative Prompt-to-App-Entwicklungsplattform, die Anfang des Jahres vorgestellt wurde, hat offiziell die Beta-Phase verlassen und ist für alle Nutzer verfügbar. Dieses bahnbrechende
Kommentare (21)
0/200
Figma veröffentlicht AI-gestütztes App Builder Tool für alle Benutzer
Figma Make, die innovative Prompt-to-App-Entwicklungsplattform, die Anfang des Jahres vorgestellt wurde, hat offiziell die Beta-Phase verlassen und ist für alle Nutzer verfügbar. Dieses bahnbrechende
Kommentare (21)
0/200
![HarryClark]() HarryClark
HarryClark
 21. August 2025 09:01:19 MESZ
21. August 2025 09:01:19 MESZ
AlphaFold 3 sounds like a game-changer! Predicting molecular interactions could speed up drug discovery big time. Curious how it stacks up against traditional lab methods. 🧬


 0
0
![PeterThomas]() PeterThomas
PeterThomas
 17. August 2025 01:01:00 MESZ
17. August 2025 01:01:00 MESZ
AlphaFold 3 sounds like a game-changer! Predicting molecular interactions with such precision could really speed up drug discovery. I'm curious how it stacks up against traditional lab methods in terms of cost and time. Anyone know? 🤔


 0
0
![JoseLewis]() JoseLewis
JoseLewis
 14. August 2025 13:00:59 MESZ
14. August 2025 13:00:59 MESZ
AlphaFold 3 sounds like a game-changer! Predicting molecular interactions could speed up drug discovery big time. Excited to see how it shapes medical breakthroughs! 🚀


 0
0
![ElijahCollins]() ElijahCollins
ElijahCollins
 14. August 2025 05:01:00 MESZ
14. August 2025 05:01:00 MESZ
AlphaFold 3 sounds like a game-changer! Predicting molecular interactions could speed up drug discovery big time. Curious how it stacks against human researchers—will it outsmart us or just make our coffee breaks longer? 😄


 0
0
![GregorySmith]() GregorySmith
GregorySmith
 6. August 2025 04:01:00 MESZ
6. August 2025 04:01:00 MESZ
AlphaFold 3 sounds like a game-changer! Predicting molecular interactions with such precision could unlock new frontiers in medicine. But I wonder, will this tech stay accessible to all researchers, or get locked behind paywalls? 🤔


 0
0
![BruceThomas]() BruceThomas
BruceThomas
 1. August 2025 08:08:50 MESZ
1. August 2025 08:08:50 MESZ
AlphaFold 3 sounds like a game-changer for science! Predicting molecular interactions could speed up drug discovery big time. Excited to see where this goes! 🚀


 0
0

Seit der Einführung im Jahr 2020 haben über 2 Millionen Forscher Google DeepMinds AlphaFold 2 genutzt, um Proteinstrukturen vorherzusagen und Durchbrüche wie Impfstoffentwicklung und Krebsbehandlungen zu fördern. Dieses Modell löste ein Problem, das Wissenschaftler über ein halbes Jahrhundert lang beschäftigte. Doch das Team bei Google DeepMind ruhte sich nicht auf seinen Lorbeeren aus, sondern krempelte die Ärmel hoch und begann an AlphaFold 3 zu arbeiten.
Im Mai von Google DeepMind und Isomorphic Labs eingeführt, hebt AlphaFold 3 die Messlatte höher. Es sagt nicht nur die Proteinfaltung voraus, sondern prognostiziert auch die Struktur und Wechselwirkungen aller Arten von Molekülen des Lebens, einschließlich DNA, RNA und Liganden – jenen kleinen Molekülen, die an Proteine binden.
„Mit AlphaFold 2 haben wir große Fortschritte bei der Lösung des Proteinfaltungspuzzles gemacht, aber die wissenschaftliche Gemeinschaft ist zu komplexeren Themen übergegangen“, erklärt Jonas Adler, Forschungswissenschaftler bei Google DeepMind. „Forscher beschäftigen sich jetzt mit Details wie der Bindung kleiner Moleküle oder der Funktionsweise von RNA, Bereiche, in denen AlphaFold 2 nicht ausreichte. Um mit den neuesten Entwicklungen in Biologie und Chemie Schritt zu halten, brauchten wir ein Werkzeug, das alle Arten von Biomolekülen verarbeiten kann.“
„Alles“ umfasst hier Liganden, die entscheidend sind, da sie etwa die Hälfte aller Medikamente ausmachen. „Bei Isomorphic Labs sehen wir das enorme Potenzial von AlphaFold 3 für die rationale Arzneimittelentwicklung, und wir nutzen es bereits täglich“, sagt Adrian Stecula, Forschungsleiter bei Isomorphic Labs. „Wir erforschen, wie neue kleine Moleküle an neue Arzneimittelziele binden, wie Proteine mit DNA und RNA interagieren und wie chemische Veränderungen Proteinstrukturen beeinflussen – das neue Modell eröffnet all diese Möglichkeiten.“
Die Einbeziehung dieser zusätzlichen Molekültypen bedeutete, mit einer Vielzahl von Kombinationen umzugehen. „Proteine sind ziemlich einfach; es gibt nur 20 Standardaminosäuren“, bemerkt Jonas. „Aber kleine Moleküle? Die sind überall, mit unendlichen Möglichkeiten. Sie sind extrem vielfältig.“
„Es ist wie Google Maps, aber für molekulare Komplexe“, sagt Lindsay Willmore, Forschungsingenieurin bei Google DeepMind. „Auch wenn du kein Programmierer bist, kannst du einfach deine Protein-, DNA-, RNA- oder kleinen Molekülsequenzen kopieren und einfügen, auf einen Button klicken und ein paar Minuten warten. Du erhältst deine Struktur zusammen mit Konfidenzmetriken, um die Vorhersage zu bewerten.“
Um AlphaFold 3 mit einer so breiten Palette von Biomolekülen zu nutzen, erweiterte das Team die Trainingsdaten um DNA, RNA, kleine Moleküle und mehr. „Wir dachten: ‚Warum nicht auf allem in unserem Datensatz trainieren, was uns bei Proteinen geholfen hat, und sehen, wie weit wir kommen?‘“, erklärt Lindsay. „Es stellte sich heraus, dass wir ziemlich weit kamen.“
Eine wesentliche Änderung in AlphaFold 3 war im letzten Teil des Modells, das die Struktur erstellt. Während AlphaFold 2 ein komplexes, geometriebasiertes Modul verwendete, wechselte AlphaFold 3 zu einem einfacheren generativen Modell basierend auf Diffusion, ähnlich wie andere fortschrittliche Bildgenerierungsmodelle wie Imagen. Diese Änderung vereinfachte den Umgang mit den neuen Molekültypen.
Diese Umstellung brachte jedoch eine neue Herausforderung: Das Diffusionsmodell erstellte fälschlicherweise eine „geordnete“ Struktur für Proteinregionen, die tatsächlich „ungeordnet“ sind – stell dir vor, du versuchst, einen Haufen chaotischer Spaghetti in eine ordentliche Spirale zu bringen.
Also griff das Team auf AlphaFold 2 zurück, das bei der Vorhersage dieser ungeordneten Wechselwirkungen glänzt. „Wir haben diese Vorhersagen von AlphaFold 2 genutzt, um AlphaFold 3 zu trainieren und ihm beizubringen, Unordnung zu erkennen und vorherzusagen“, sagt Lindsay.
„Wir haben einen Spruch: ‚Vertraue den Fusilli, lehne die Spaghetti ab‘“, fügt Jonas lachend hinzu.
Das Team ist begeistert, wie AlphaFold 3 in Bereichen wie Genomik und Arzneimitteldesign eingesetzt wird. „Es ist erstaunlich zu sehen, wie weit wir gekommen sind“, sagt Jonas. „Was einst schwierig war, ist jetzt einfach, und was unmöglich war, ist jetzt in Reichweite. Es gibt immer noch harte Nüsse zu knacken, aber wir sind begeistert, was AlphaFold 3 uns ermöglicht.“
 Anthropic löst Rechtsstreit über AI-generierte Buchpiraterie
Anthropic hat einen bedeutenden Urheberrechtsstreit mit US-Autoren beigelegt. Das Unternehmen hat einer vorgeschlagenen Sammelklage zugestimmt, mit der ein potenziell kostspieliger Prozess vermieden w
Anthropic löst Rechtsstreit über AI-generierte Buchpiraterie
Anthropic hat einen bedeutenden Urheberrechtsstreit mit US-Autoren beigelegt. Das Unternehmen hat einer vorgeschlagenen Sammelklage zugestimmt, mit der ein potenziell kostspieliger Prozess vermieden w
 Figma veröffentlicht AI-gestütztes App Builder Tool für alle Benutzer
Figma Make, die innovative Prompt-to-App-Entwicklungsplattform, die Anfang des Jahres vorgestellt wurde, hat offiziell die Beta-Phase verlassen und ist für alle Nutzer verfügbar. Dieses bahnbrechende
Figma veröffentlicht AI-gestütztes App Builder Tool für alle Benutzer
Figma Make, die innovative Prompt-to-App-Entwicklungsplattform, die Anfang des Jahres vorgestellt wurde, hat offiziell die Beta-Phase verlassen und ist für alle Nutzer verfügbar. Dieses bahnbrechende
 21. August 2025 09:01:19 MESZ
21. August 2025 09:01:19 MESZ
AlphaFold 3 sounds like a game-changer! Predicting molecular interactions could speed up drug discovery big time. Curious how it stacks up against traditional lab methods. 🧬


 0
0
 17. August 2025 01:01:00 MESZ
17. August 2025 01:01:00 MESZ
AlphaFold 3 sounds like a game-changer! Predicting molecular interactions with such precision could really speed up drug discovery. I'm curious how it stacks up against traditional lab methods in terms of cost and time. Anyone know? 🤔


 0
0
 14. August 2025 13:00:59 MESZ
14. August 2025 13:00:59 MESZ
AlphaFold 3 sounds like a game-changer! Predicting molecular interactions could speed up drug discovery big time. Excited to see how it shapes medical breakthroughs! 🚀


 0
0
 14. August 2025 05:01:00 MESZ
14. August 2025 05:01:00 MESZ
AlphaFold 3 sounds like a game-changer! Predicting molecular interactions could speed up drug discovery big time. Curious how it stacks against human researchers—will it outsmart us or just make our coffee breaks longer? 😄


 0
0
 6. August 2025 04:01:00 MESZ
6. August 2025 04:01:00 MESZ
AlphaFold 3 sounds like a game-changer! Predicting molecular interactions with such precision could unlock new frontiers in medicine. But I wonder, will this tech stay accessible to all researchers, or get locked behind paywalls? 🤔


 0
0
 1. August 2025 08:08:50 MESZ
1. August 2025 08:08:50 MESZ
AlphaFold 3 sounds like a game-changer for science! Predicting molecular interactions could speed up drug discovery big time. Excited to see where this goes! 🚀


 0
0